
除了字典中包含的由 注释的首字母缩写词之外,还可以在每个文档的基础上检测到新颖的首字母缩写词声明。当检测到首字母缩写词定义(形式为“物种(首字母缩略词)”时,其中物种在字典中,首字母缩写词是大写字母、数字或连字符的序列),该首字母缩写词的所有后续出现也会在文档中标记。
删除常用英语单词
基于一个简单的物种名称列表,这些物种名称在不提及物种时通常出现在英语中(参见附加文件3),我们删除了列表中包含物种术语组合的任何提及 . 这消除了“spot”(对于 )和“permit”(对于 )等同义词,并大大减少了系统产生的误报数量。
为模棱两可的提及分配概率
最后,任何仍然模棱两可的提及都被分配了提及特定物种的可能性的概率。模糊提及的概率基于所有 MEDLINE 和 PubMed Central 全文文档的开放访问子集中所涉及物种的明确提及的相对频率。首字母缩写词的概率基于 检测到的首字母缩写词定义的相对频率(见上文)。例如,对于模棱两可的提及“C. elegans”,出现秀丽隐杆线虫的概率会非常高,而出现Crella elegans的概率会很高会低很多。对于首字母缩略词“HIV”(可能同时指“人类免疫缺陷病毒”,更不常见的是“希波克拉底无关变量”),它指代“人类免疫缺陷病毒”的可能性非常高。
这些概率启用了另一种启发式消歧形式:在模棱两可的提及具有高于给定截止值(例如 99%)的概率的物种替代的情况下,提及可以完全消除该物种的歧义(例如术语“C. elegans”可以被消除为 elegans)。同样,如果所有与物种相关的提及概率之和小于给定阈值(例如 1%),则可以删除提及;这可能发生在首字母缩略词中,在 99% 以上的情况下,首字母缩略词用于非物种术语。这些级别在准确性和模糊性最小化之间进行了权衡,并且可以在标记后根据用户的个人需求进行调整。
输入和输出格式
能够处理各种文档 XML 格式,包括 MEDLINE XML、PMC XML、Biomed Central XML和 Open Text Mining XML。此外,它还可以处理来自本地存储文件和远程数据库服务器的纯文本文档。物种名称识别结果可以存储到基于对峙的制表符分隔值文件、XML 文档、HTML 文档(用于结果的简单可视化)和远程 MySQL 数据库表中。
用于物种标记的文档集
在整个工作中,使用了三个不同的文档集来识别和规范物种名称。对于所有集合,2008 年之后发布的任何文档都被删除,以创建固定和可重复的文档集合,并避免在项目过程中因数据库记录更新而可能出现的差异。
医疗线
MEDLINE 是 PubMed 文章摘要的主要数据库,包含超过 1800 万条条目。然而,许多条目实际上并不包含任何摘要。如果仅计算截至 2008 年底发表的包含摘要的条目,则文件数量刚刚超过 990 万份。
PubMed Central 开放获取子集
PMC 免费提供超过一百万篇全文文章。不幸的是,其中只有大约 10%(截至 2008 年底发布了 105,106 篇)是真正的开放访问并可用于不受限制的文本挖掘。此 PMC 的开放存取 (OA) 子集中的文章在此称为“PMC OA”。PMC OA 中的大部分文章都是基于 XML 文件,但有些是通过扫描非数字文章(29,036 个文档)的光学字符识别(OCR)创建的,还有一些是通过转换便携式文档格式(PDF ) 文档到文本(9,287 个文档)。我们注意到,对于使用 OCR 或 pdf 到文本软件生成的 PMC OA 文档,不会从这些文档中删除参考。正因为如此,出现在参考标题中的物种名称可能会被标记。对于所有其他文件(MEDLINE、即不处理参考标题)。
PMC OA 的摘要
PMC OA 集中所有文章的摘要形成一个称为“PMC OA abs”的集。PMC OA 摘要是从 PMC OA XML 文件的摘要部分获得的,或者如果 XML 文件中不存在这样的部分,则从相应的 MEDLINE 条目获得(当文章是通过 OCR 或 pdf 到文本工具生成时会发生这种情况) . PMC OA 摘要包含 88,962 篇文档,明显少于 PMC OA 中的文档数量(105,106 篇)。这是因为并非所有 PMC 文章都被 MEDLINE 索引,因此一些 OCR 或 pdf 转文本文档没有对应的 MEDLINE 条目,使得准确提取摘要不可行。在 88,962 篇摘要中,有 65,739 篇(74%)是从 XML 文档中提取的,其余部分是从相应的 MEDLINE 文档中提取的。
PMC OA 全文文档集的划分
如上一节所述,不可能可靠地提取 PubMed Central 中大约五分之一的全文文章的摘要,因为它们在 PMC XML 或相应的 MEDLINE 条目中没有摘要部分。我们选择不从我们的分析中删除这些全文文章,因为它们包含 PubMed Central 中的大量文档子集,并且它们的排除可能会使我们的结果产生偏差。但是,它们的包含使得基于 PMC OA 摘要和所有 PMC OA 全文文档的结果的直接比较变得困难,因为 PMC OA 全文集中存在一些文档,而 PMC OA 摘要集中缺少这些文档。为了在文档层面解决这个问题,我们创建了“PMC OA full (abs)”集,其中包含可以提取摘要的 88,962 个全文文档,允许直接比较完全相同文章的全文文档和摘要。不幸的是,该文档集仍然不允许在摘要和全文之间进行直接提及级别的比较,因为来自 MEDLINE 条目的偏移坐标和 PMC OA 全文文档不兼容。因此,我们创建了“PMC OA full (xml)”集,该集仅包含 65,739 个全文文档,其中可以从相应的 PMC XML 文件中提取摘要。使用此 PMC OA 全文 XML 集,还可以在相同偏移坐标上对相同文档集执行提及级别比较。我们注意到“PMC OA”是指完整的 105,106 个全文文档集,我们也可以将其表示为“PMC OA full (all)”。
用于评估的文档集
目前,不存在专门针对物种提及进行注释的生物医学文档的开放访问语料库。因此,我们创建了许多自动生成的评估集,以分析 和其他物种名称标记软件的准确性。由于它们所基于的数据的性质,许多这些评估集只能在文档级别进行分析。此外,这些自动生成的评估集都不是基于专门为注释物种提及而创建的数据。正因为如此,我们创建了一个为物种提及手动注释的全文文章的评估集。每个评估集覆盖的文档、物种和标签的数量如表1所示完整的手动注释文档可以在项目网页上找到。
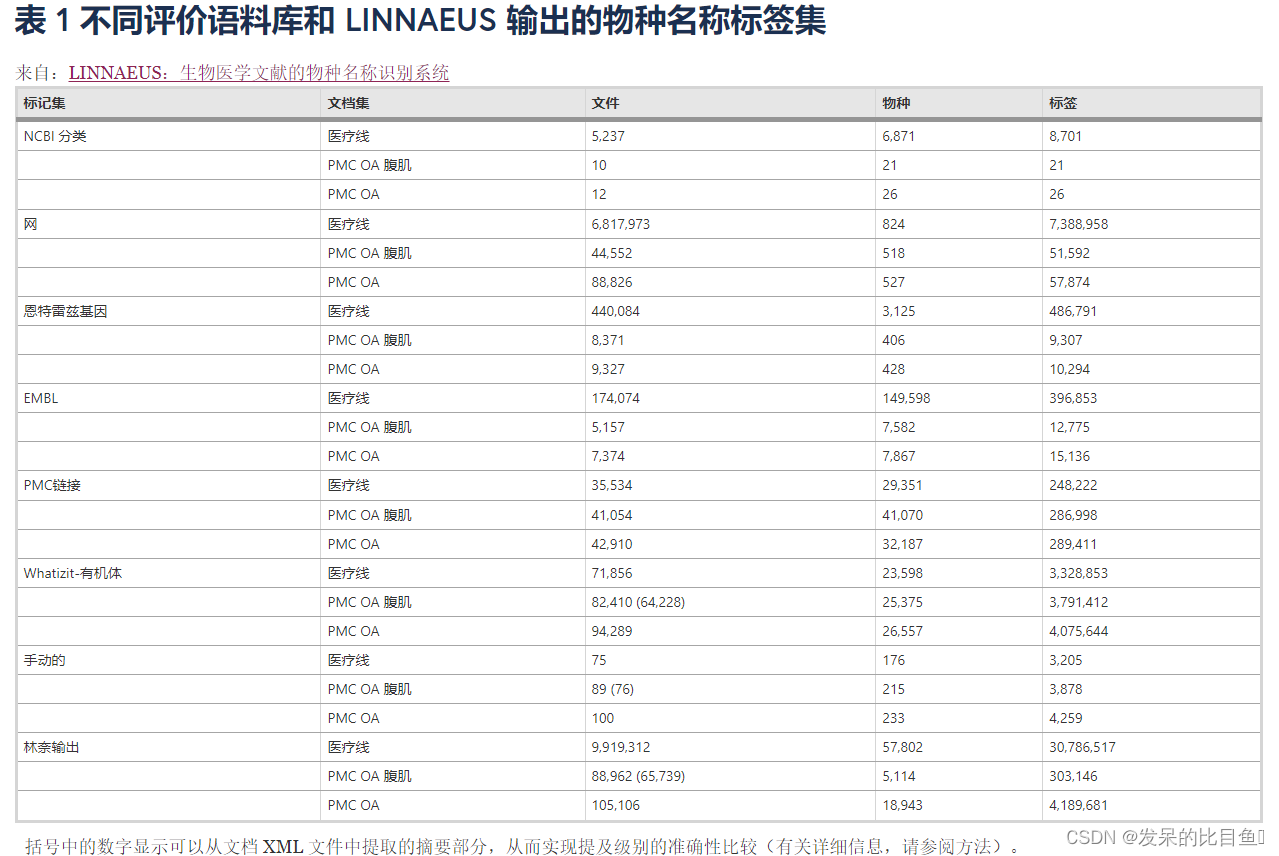
NCBI 分类引文

NCBI 分类中的一些物种条目包含对讨论该物种的研究文章的引用。对于这些文件,我们假设该物种最有可能在文章的某处被提及,从而使相对回忆成为一种有用的衡量标准。NCBI 分类引文于 2009 年 6 月 1 日下载。
医学主题词条
MEDLINE 中的每篇文章都有相关的 MeSH 术语,指定文章中讨论的主题。这些术语的一个子集与物种有关,并且可以通过统一医学语言系统 (UMLS) 映射到 NCBI 分类物种条目。然而,由 MeSH 术语表示的物种数量是有限的。总共只有 1,283 个物种的 MeSH 术语,在 MEDLINE 的 MeSH 标签中实际出现的物种只有 824 个。此外,赋予文章的 MeSH 术语并不能保证该术语在文档中明确提及。此外,预计文档中提及的总物种中只有一小部分会在 MeSH 标签中表示(只有所谓的焦点物种),导致使用该语料库的精度估计不如召回信息量大。
Entrez 基因条目
MEDLINE 中的每篇文章都有相关的 MeSH 术语,指定文章中讨论的主题。这些术语的一个子集与物种有关,并且可以通过统一医学语言系统 (UMLS) 映射到 NCBI 分类物种条目。然而,由 MeSH 术语表示的物种数量是有限的。总共只有 1,283 个物种的 MeSH 术语,在 MEDLINE 的 MeSH 标签中实际出现的物种只有 824 个。此外,赋予文章的 MeSH 术语并不能保证该术语在文档中明确提及。此外,预计文档中提及的总物种中只有一小部分会在 MeSH 标签中表示(只有所谓的焦点物种),导致使用该语料库的精度估计不如召回信息量大。
EMBL 记录
与 Entrez 基因记录类似,许多 EMBL序列记录还包含有关该序列来自哪个物种以及该序列是在哪篇文章中报道的信息。假设在报告核苷酸序列的论文中明确提到了物种,这可以提取物种-文章映射。然而,与 Entrez 基因集一样,这并不能保证,除了具有报告序列的物种之外,讨论的任何物种都不会出现在评估集中(再次导致精确测量无信息)。该评估集使用了 EMBL 的 r98 版本。
PubMed 中央链接
尽管没有在任何出版物中描述,NCBI 对 PMC 中包含的全文文章进行物种识别文本挖掘。这些分类“链接”可以在查看 PMC 上的文章时访问,也可以通过 NCBI e-utils Web 服务下载。通过下载这些链接,可以创建与召回率和精度相关的评估集(尽管仅在文档级别)。PMC 链接数据于 2009 年 6 月 1 日下载。
为了评估提及级别的准确性并将 与另一个物种名称识别系统进行基准比较,PMC OA 集中的所有文档都通过 Web 服务管道发送。不幸的是, Web 服务无法处理大约 10% 的 PMC OA 文档(参见表1),因此无法进行比较。 标记于 2009 年 6 月 25 日执行。
人工标注的金标准语料库
由于所有前面描述的评估集都受到它们没有专门为物种名称注释的事实的限制,因此很明显需要这样一个集来测量 的真实准确性。因为没有这样的评估集可用,所以从 PMC OA 文档集中随机选择了 100 个全文文档并为物种提及进行了注释。由于这项工作的重点是物种而不是属或其他更高阶的分类单位,因此语料库仅针对物种进行了注释(除了在提及物种时错误地使用了属名的情况)。
所有提及的物种术语均手动注释并标准化为预期物种的 NCBI 分类 ID,但作者未提及该物种的术语除外。一个常见的例子是“Fisher 精确检验”(“Fisher”是Martes 的同义词,但在这种情况下指的是发明统计检验的 Ronald Aylmer Fisher 爵士)。在 NCBI 分类中不存在物种 ID 的情况下(主要发生在特定物种菌株中),它们的物种 ID 为 0(在 NCBI 分类中不使用)。
带注释的提及也被分配到以下类别,这些类别表明提及的特定特征,可用于评估分析:
(一)词汇类别:
提及可能属于多个类别(例如,它可能既用作修饰符又可能拼写错误),或者根本不属于任何类别(即只是普通提及,这是最常见的情况)。表2显示了与每个类别相关的物种标签数量的摘要。这些类别可以深入了解物种名称在文献中拼写错误或使用不正确的频率。它们还可以对 或针对该语料库评估的任何其他软件所做的任何预测错误进行更深入的分析。在该语料库中注释的 4259 个物种中,72% (3065) 是常用名称,这加强了在处理生物医学研究文章时能够准确识别常用名称的重要性。
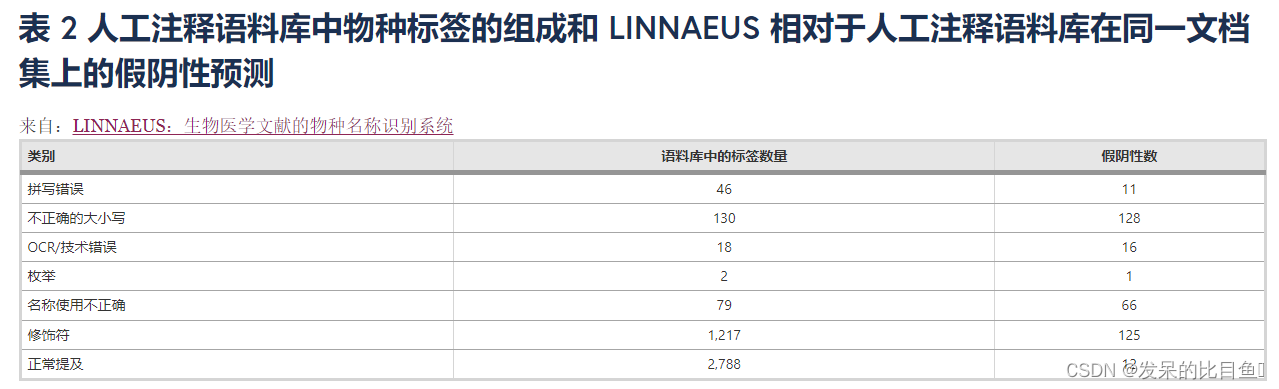
为了估计手动注释的可靠性,10% 的语料库(10 个文档)也由第二个注释器注释,并计算了注释器间协议 (IAA)。总共有 406 个物种提及在 10 个文件中由至少一个注释者注释。在这 406 次提及中,368 次被两个注释器(提及位置和物种标识符)相同地注释。Cohen 对注释者间一致性的 k 度量 [ 53 ] 计算为 k = 0.89。IAA 分析的详细信息可在附加文件4中找到。
绩效评估
将 生产的标签与评估参考集中的标签进行比较,以确定系统的性能。如果特定标签同时出现在 集和参考集中,则称为真阳性(TP);如果它仅出现在 集中,则称为误报 (FP);如果它仅出现在参考集中,则称为假阴性(FN)。这在文档级别(不考虑文档中标签的位置)和提及级别(标签位置必须完全匹配)上执行。对于信息仅在文档级别可用的评估集,不执行提及级别评估。在不明确提及的情况下,如果提及至少包含“真实”物种,则该提及被视为 TP(并且,对于提及水平分析,位置正确)。我们注意到 试图识别文件中提到的所有物种,因此报告的物种数量没有限制。
结果
我们将 系统应用于 2008 年或之前发表的近 1000 万篇 MEDLINE 摘要和超过 100,000 篇 PMC OA 文章(表1)。使用四个 Intel Xeon 3 GHz CPU 内核和 4 GB 内存,MEDLINE 的文档集标记大约需要 5 小时,PMC OA 摘要需要 2.5 小时,PMC OA 需要 4 小时。(我们注意到影响处理时间的主要因素是 Java XML 文档解析而不是实际的物种名称标记。)这些物种标记实验远远超过了任何先前报告的规模,并代表了文本挖掘在整个 PMC OA 语料库中的第一个应用。在 MEDLINE 中检测到超过 57,000 个不同物种的超过 3000 万个物种标签,在 PMC OA 中检测到近 19,000 个物种的超过 400 万个物种标签。 在 74% 的 MEDLINE 文章、72% 的 PMC OA 摘要和 96% 的 PMC OA 全文文章中识别出物种。从NCBI分类词典中的物种总数来看,15%的NCBI词典中的物种被在MEDLINE中找到,1.3%在PMC OA摘要中找到,4.9%在PMC OA全文中找到文章。MEDLINE 或 PMC OA 摘要中的物种名称密度分别比 PMC OA 全文文章低 30 倍和 3 倍;相对于全文文档,两组摘要中物种提及的密度都低 11 倍。
MEDLINE 和 PubMed Central 中提到的物种的歧义
在所有 MEDLINE 和 PMC OA 中,11-14% 的物种提及是模棱两可的。因此,物种名称歧义的水平与基因名称中的跨物种歧义处于相同的顺序,并表明某种形式的消歧对于准确的物种名称规范化是必要的。表3显示了 消歧步骤之前和之后的标记文档集的歧义级别. 歧义级别的计算方法是歧义提及的数量除以提及的总数,其中当提及映射到多个物种时,会计算歧义提及。消歧方法“无”显示任何消歧之前的值;“earlier”通过扫描文档中较早的明确提及来消除歧义,为了比较,“whole”通过扫描整个文档中的明确提及来消除歧义。“严格”消歧不考虑正确物种提及的相关概率,而“近似”表示对单个物种具有高于 99% 概率或所有物种概率之和低于 1% 的任何提及的消歧。
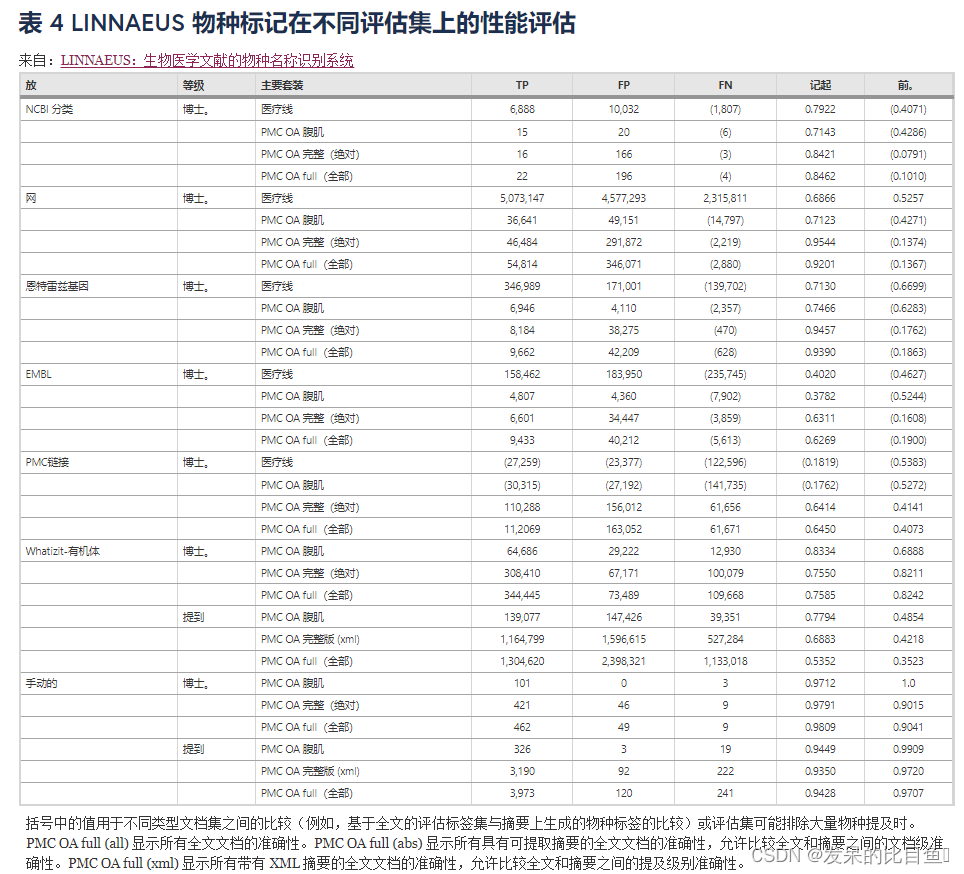
评估 物种名称标记
与评估集中的物种相比, 发现的物种提及的评估如表4所示. 对于文档级评估集(NCBI 分类参考、MeSH 标签、Entrez 基因参考、EMBL 参考和 PMC 链接),文档级标签直接与 在 MEDLINE、PMC OA 摘要或 PMC OA 中找到的标签进行比较文件。对于提及级评估集( 输出和手动注释集),仅在评估集和 PMC OA XML 之间直接比较标签,因为 PMC OA XML 是唯一与评估集在相同偏移坐标上的文档集(见方法)。对于自动生成的集合,我们在评估集中如何注释物种的背景下解释召回和精度,以提供对假阳性和假阴性的定性分析。对于人工标注的金标准评估集,
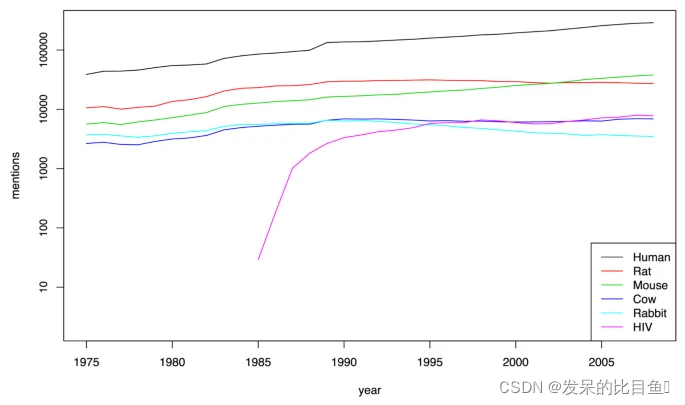
讨论
物种名称识别和规范化越来越被认为是文本挖掘和生物信息学中的一个重要主题,不仅因为它可以为最终用户提供直接优势,而且还可以指导其他软件系统。虽然之前已经报道了许多执行物种名称识别和/或科学名称和同义词标准化的工具,这里介绍的工作以多种独特的方式为该领域做出了贡献。其中包括强大的、开源的、独立的应用程序的可用性(其他工具要么不公开提供,只能作为 Web 服务提供,要么不能识别常用名称)、物种标记的规模(所有 MEDLINE 和 PMC OA 直到2008)、评估的深度和严谨性(其他工具不针对规范化的数据库标识符进行评估,或者仅限于少量文档样本)和准确性(与其他可用工具相比, 表现出更好的性能,主要是由于更好地处理含糊不清的提及和包含其他同义词)。此外,我们提供第一个开放访问,
评估物种名称识别软件需要人工注释的金标准
任何生物信息学应用程序的相对性能仅与与之比较的评估集一样好。在物种名称识别软件的情况下,在当前工作之前,没有开放访问的生物医学文本中物种名称注释的手动注释数据集作为评估的黄金标准。在这个项目中,我们研究了四种不同的自动生成的评估集(NCBI 分类引文、MeSH 标签、Entrez 基因参考、EMBL 引文),这些评估集基于策展的文档-物种对。我们还根据使用文本挖掘软件(PMC 和 )预测的文档物种对研究了两个不同的自动生成的评估集。尽管当文档集和评估集属于同一类型时,可以解释 的召回(例如全文),由于在任何这些评估集中对物种提及的不完整或不完善的注释,我们的系统的精度无法准确评估。我们得出结论,从“次要”来源(例如文档基因(例如Entrez 基因)或文档序列(例如EMBL)映射)自动推断出的文档-物种映射评估集在评估物种名称识别软件中的价值有限。
由于自动生成的评估集的固有局限性(包括物种名称的不完整注释或不正确的消歧),因此创建了手动注释的评估语料库。对手动注释评估语料库的评估显示, 的性能非常好,在提及级别上具有 94.3% 的召回率和 97.1% 的准确率,在文档级别上具有 98.1% 的召回率和 90.4% 的准确率。没有一个自动生成的评估集能接近揭示使用 进行物种名称识别的这种精度水平。这些结果强调了我们手动注释的黄金标准评估集的重要性,并建议在自动生成的评估集上评估其他系统可能低估了系统精度。拥有高质量评估集的一个有趣观察是,召回率高于文档级别的准确率,而准确率高于提及级别的召回率。造成这种情况的一个原因是,当作者使用非标准或拼写错误的名称时,他们通常会在整个文档中多次使用这些名称,导致在提及级别上出现多个误报,但仅在文档级别上出现一次。相反,误报在文档中更分散,导致提及和文档级别评估的误报计数差异很小。
提高全文文章中物种名称识别的准确性
目前绝大多数文本挖掘研究都是针对生物医学文章的摘要进行的,因为它们在 PubMed 中免费提供,分析所需的计算资源较少,并且被认为包含最高密度的信息。然而,越来越多的证据表明,全文文章的信息检索效果更好,因为生物医学术语的覆盖率高于摘要。我们的物种名称识别结果支持这一结论,对于大多数测试的评估集,全文文章的物种名称召回率高于摘要(表4) 并且几乎所有 (96%) 全文文章都被标记为至少一个物种名称。对全文文章进行术语识别的好处在物种名称的情况下可能特别有用,因为与疾病、基因或化学品和药物的术语相比,生物术语在生物医学文档的不同部分中的分布似乎更加统一。
我们的结果还清楚地表明,通过搜索明确提及来消除物种提及的歧义在全文文章中比在摘要中更成功。因此,正如之前发现的基因名称,全文覆盖率的增加对物种名称消歧有额外的好处,因为在处理全文文章时,消歧算法可以获得更多信息。有趣的是,我们发现无论是在文本的前面还是在整个文本中扫描明确提及,歧义的程度都会下降,这可能是因为文章的材料和方法部分通常位于论文的末尾。在搜索明确提及后,我们发现生物医学文本中物种名称的歧义水平很低(3-5%),如果可以容忍少量错误,可以使用概率方法进一步降低(1-3%)。
结论
我们开发并评估了一个强大的开源软件系统 ,它可以快速准确地识别生物医学文件中的物种名称,并将它们规范化为 NCBI 分类中的标识符。 系统的低歧义性、高召回率和高精度使其非常适合生物医学文本中的自动物种名称识别。生物医学领域的 物种识别可以通过包含细胞系名称来增强 [ 67 ],这些名称通常充当产生它们的物种的生物代理。 也可能在其他问题领域表现良好,例如生态学和分类学文献,前提是提供高质量的物种名称词典(例如 [ 68]),尽管这仍然是未来研究的开放领域。进一步开发 以在生物医学文献之外更广泛地应用可能需要与其他方法集成,例如基于规则的物种名称识别系统(例如 ),我们目前的目标是在未来提供此类方法的实现,以便能够使用 提供的文件处理方法。 的可用性现在为在文本中使用物种名称的下游应用程序提供了机会,包括将物种名称集成到更大的生物信息学管道中,生物医学文本中物种名称的语义标记,以及跨物种名称使用趋势的数据挖掘文件和时间。
*请认真填写需求信息,我们会在24小时内与您取得联系。